クラスタリング解析は解るような、解らないような、知識がない方に説明が難しい「デンドログラムをカッコよく描写」する方法を考えがちです。でも、ふと「デンドログラムの描写から解釈を導き出す」表現方法はないかと考える場合があります。
そんな欲求を満たすことができるかもしれない「hybridHclust」パッケージを紹介します。
クラスタリング解析を数学的に理解したい方には「Rで学ぶクラスタ解析」を紹介します。読みやすく、大変面白い本でした。
パッケージのバージョンは1.0-5。R version 3.2.1でコマンドを確認しています。
パッケージのインストール
下記コマンドを実行してください。
#パッケージのインストール
install.packages("hybridHclust")実行コマンドの紹介
詳細はコメント、パッケージヘルプを確認してください。
#パッケージの読み込み
library("hybridHclust")
library("RColorBrewer")
#RColorBrewerパッケージのインストール
#install.packages("RColorBrewer")
#デンドログラムの計算:eisenClusterコマンド
#methodオプション:“euclidean”, “squared.euclidean”, “correlation”, “uncentered.correlation”
#データ例の作成
set.seed(99)
TestData <- matrix(rnorm(500), 5, 100)
TestData <- rbind(TestData, TestData[rep(1, 4),] + matrix(rnorm(400), 4, 100))
TestData <- rbind(TestData, TestData[2:5,] + matrix(rnorm(400) , 4, 100))
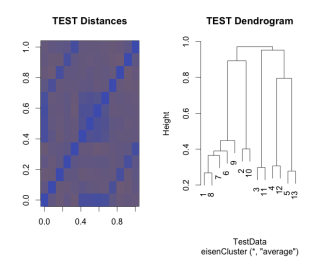
EC <- eisenCluster(TestData, method = "correlation")
#描写領域の分割
par(mfrow = c(1, 2))
#ヒートマップの描写
image(1-cor(t(TestData)), main = "TEST Distances",
zlim = c(0,2), col = colorRampPalette(c("#4b61ba", "#a87963"))(100))
#デンドログラムの描写
plot(EC, main = "TEST Dendrogram")
#相互クラスタの計算と描写:mutualClusterコマンド
#データ例の作成
TestData2 <- cbind(c(-1.4806,1.5772,-0.9567,-0.92,-1.9976,-0.2723,-0.3153),
c(-0.6283,-0.1065,0.428,-0.7777,-1.2939,-0.7796,0.012))
#描写領域の分割
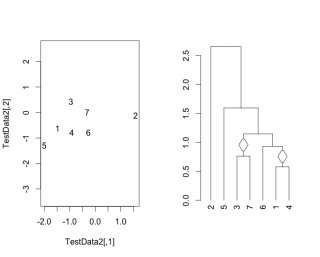
par(mfrow = c(1,2))
#データ例を描写
plot(TestData2, pch = as.character(1:nrow(TestData2)), asp = 1)
#デンドログラムの描写
mutualCluster(TestData2, plot = TRUE)
#相互クラスタの距離を表示:get.distancesコマンド
get.distances(mutualCluster(TestData2, plot = FALSE))
[[1]]
7
3 0.7644933
[[2]]
4
1 0.5801661出力例
・eisenClusterコマンド
・mutualClusterコマンド
少しでも、あなたのウェブや実験の解析が楽になりますように!!