データのlong型やwide型への変形、欠損値やネスト構造化に便利な「tidyr」パッケージのコマンドを再確認しました。「gather」コマンドや「spread」コマンドが有名なパッケージです。
パッケージバージョンは0.7.1。windows 10のR version 3.4.1で動作を確認しています。
パッケージのインストール
下記コマンドを実行してください。
#パッケージのインストール
install.packages("tidyr")パッケージの読み込みとデータ例の作成
データ例「TestData」を利用しコマンドを紹介しています。
#パッケージの読み込み
library("tidyr")
###データ例の作成#####
n <- 5
set.seed(10)
TestData <- data.frame(Group = I(sample(c(paste0("Group", 1:2), NA),
n, replace = TRUE)),
Data1 = rnorm(n),
Data2 = rnorm(n) + rnorm(n) + rnorm(n),
Data3 = sample(0:1, n, replace = TRUE),
Data4 = sample(LETTERS[1:26], n, replace = TRUE))
#確認
TestDataパイプ演算子「%>%」の紹介
パイプ演算子の左側のオブジェクトをパイプ演算子右側の関数の最初の引数へ渡します。利用することでコマンド記述が文法的になり内容の理解が捗ります。でもコメントの記述は大事です。
#TestData[1:2, 1]を三回繰り返す
#%>%を使わない
rep(x = TestData[1:2, 1], time = 3)
[1] "Group2" "Group1" "Group2" "Group1" "Group2" "Group1"
#%>%を使う
TestData[1:2, 1] %>% rep(time = 3)
[1] "Group2" "Group1" "Group2" "Group1" "Group2" "Group1"データ変形コマンド
データをlong型に変形する「gather」コマンド、long型データをwide型に変形する「spread」コマンド、データをネスト構造にする「nest」コマンド、ネスト構造からデータを取り出す「unnest」コマンドの紹介です。
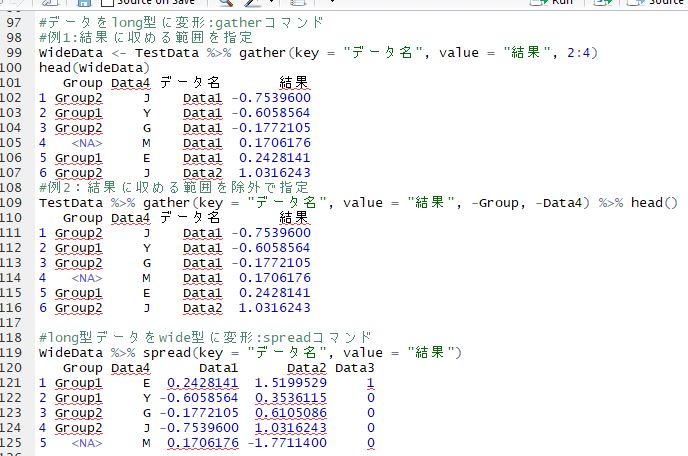
#データをlong型に変形:gatherコマンド
#例1:結果に収める範囲を指定
WideData <- TestData %>% gather(key = "データ名", value = "結果", 2:4)
head(WideData)
Group Data4 データ名 結果
1 Group2 J Data1 -0.7539600
2 Group1 Y Data1 -0.6058564
3 Group2 G Data1 -0.1772105
4 <NA> M Data1 0.1706176
5 Group1 E Data1 0.2428141
6 Group2 J Data2 1.0316243
#例2:結果に収める範囲を除外で指定
TestData %>% gather(key = "データ名", value = "結果",
-Group, -Data4) %>% head()
Group Data4 データ名 結果
1 Group2 J Data1 -0.7539600
2 Group1 Y Data1 -0.6058564
3 Group2 G Data1 -0.1772105
4 <NA> M Data1 0.1706176
5 Group1 E Data1 0.2428141
6 Group2 J Data2 1.0316243
#long型データをwide型に変形:spreadコマンド
WideData %>% spread(key = "データ名", value = "結果")
Group Data4 Data1 Data2 Data3
1 Group1 E 0.2428141 1.5199529 1
2 Group1 Y -0.6058564 0.3536115 0
3 Group2 G -0.1772105 0.6105086 0
4 Group2 J -0.7539600 1.0316243 0
#範囲に対するdata.frameをlistへ保存(ネスト構造化):nestコマンド
#範囲の指定はgatherコマンドと同じ
NestData <- TestData %>% gather(key = "データ名",
value = "結果", -Group, -Data4) %>%
nest(-Group, .key = "list")
#データを取り出す
NestData[2, 2]
[[1]]
Data4 データ名 結果
2 Y Data1 -0.6058564
5 E Data1 0.2428141
7 Y Data2 0.3536115
10 E Data2 1.5199529
12 Y Data3 0.0000000
15 E Data3 1.0000000
#ネスト構造からデータを取り出す:unnestコマンド
unnest(NestData, .id = "List_No.")
Group List_No. Data4 データ名 結果
1 Group2 1 J Data1 -0.7539600
2 Group2 1 G Data1 -0.1772105
3 Group2 1 J Data2 1.0316243
4 Group2 1 G Data2 0.6105086
5 Group2 1 J Data3 0.0000000
6 Group2 1 G Data3 0.0000000損値とデータ組み合わせのコマンド
欠損値を含むデータを削除する「drop_na」コマンド、欠損値を直前に出現した値で保管する「fill」コマンド、欠損値を置換する「replace_na」コマンド、指定した列の全てを組み合わせる「expand」コマンド、存在するデータを組み合わせる「nesting」コマンド、指定したベクトルと行を全て組み合わせる「crossing」コマンドの紹介です。
#欠損値を含むデータを削除:drop_naコマンド
TestData %>% drop_na()
Group Data1 Data2 Data3 Data4
1 Group2 -0.7539600 1.0316243 0 J
2 Group1 -0.6058564 0.3536115 0 Y
3 Group2 -0.1772105 0.6105086 0 G
5 Group1 0.2428141 1.5199529 1 E
#欠損値を直前に出現した値で保管:fillコマンド
#昇降の設定:.directionオプション;"down","up"
TestData %>% fill(Group, .direction = "down")
Group Data1 Data2 Data3 Data4
1 Group2 -0.7539600 1.0316243 0 J
2 Group1 -0.6058564 0.3536115 0 Y
3 Group2 -0.1772105 0.6105086 0 G
4 Group2 0.1706176 -1.7711400 0 M
5 Group1 0.2428141 1.5199529 1 E
#欠損値を置換:replace_naコマンド
#factorには適応できない
TestData %>% replace_na(list(Group = 3))
Group Data1 Data2 Data3 Data4
1 Group2 -0.7539600 1.0316243 0 J
2 Group1 -0.6058564 0.3536115 0 Y
3 Group2 -0.1772105 0.6105086 0 G
4 3 0.1706176 -1.7711400 0 M
5 Group1 0.2428141 1.5199529 1 E
#指定した列の全てを組み合わせる:expandコマンド
TestData %>% expand(Data3, Group)
# A tibble: 6 x 2
Data3 Group
<int> <chr>
1 0 Group1
2 0 Group2
3 1 Group1
4 1 Group2
#存在するデータを組み合わせる:nestingコマンド
#expandコマンドと組み合わせると便利
TestData %>% expand(nesting(Data3, Group))
# A tibble: 4 x 2
Data3 Group
1 0 Group1
2 0 Group2
#指定したベクトルと行を全て組み合わせる:crossingコマンド
#直積集合
TestData %>% crossing(1:2, c("A", "B")) %>% head()
Group Data1 Data2 Data3 Data4 1:2 c("A", "B")
1 Group2 -0.7539600 1.0316243 0 J 1 A
2 Group2 -0.7539600 1.0316243 0 J 1 B
3 Group2 -0.7539600 1.0316243 0 J 2 A
4 Group2 -0.7539600 1.0316243 0 J 2 B
5 Group1 -0.6058564 0.3536115 0 Y 1 A
6 Group1 -0.6058564 0.3536115 0 Y 1 B結合と分解のコマンド
列を結合する「unite」コマンド、データ内容を列に分割する「separate」コマンド、列データを行方向に分割する「separate_rows」コマンド
#列を結合:uniteコマンド
UniteData <- TestData %>% unite(UniteData, c(Data3, Data4),
sep = "_", remove = FALSE)
#確認
UniteData
Group Data1 Data2 UniteData Data3 Data4
1 Group2 -0.7539600 1.0316243 0_J 0 J
2 Group1 -0.6058564 0.3536115 0_Y 0 Y
3 Group2 -0.1772105 0.6105086 0_G 0 G
#データ内容を列に分割:separateコマンド
UniteData %>% separate(UniteData, into = c("Data3_1", "Data4_1"),
sep = "_", remove = FALSE)
Group Data1 Data2 UniteData Data3_1 Data4_1 Data3 Data4
1 Group2 -0.7539600 1.0316243 0_J 0 J 0 J
2 Group1 -0.6058564 0.3536115 0_Y 0 Y 0 Y
3 Group2 -0.1772105 0.6105086 0_G 0 G 0 G
#列データを行方向に分割:separate_rowsコマンド
UniteData %>% separate_rows(UniteData, sep = "_", convert = TRUE)
Group Data1 Data2 Data3 Data4 UniteData
1 Group2 -0.7539600 1.0316243 0 J 0
2 Group2 -0.7539600 1.0316243 0 J J
3 Group1 -0.6058564 0.3536115 0 Y 0
4 Group1 -0.6058564 0.3536115 0 Y Y
5 Group2 -0.1772105 0.6105086 0 G 0
6 Group2 -0.1772105 0.6105086 0 G G少しでも、あなたの解析が楽になりますように!!