デンドログラム描写には欠かせないパッケージ「dendextend」です。本パッケージがあればデンドログラムの描写には困らないと思います。Rパッケージ付属のdistコマンドでは距離の計算に”pearson”が使用できないので、「amap」パッケージをインストールしています。
パッケージのバージョンは1.15.2。実行コマンドはwindows 11のR version 4.1.2で確認しています。
パッケージのインストール
下記コマンドを実行してください。
#パッケージのインストール
install.packages("dendextend")
install.packages("dendextendRcpp")
install.packages("amap")実行コマンド
下記コマンドを実行してください。詳細はコメントを確認してください。その他コマンドはパッケージヘルプを確認してください。
#パッケージの読み込み
library("dendextend")
library("dendextendRcpp")
library("amap")
#####準備#####################
#データ例の作成
#25*4のデータ
TestData <- matrix(sample(1:100, 100, replace = TRUE), 25, 4)
TestData <- data.frame(row.names = paste0("TEST", 1:25), TestData)
colnames(TestData) <- paste0("Data", 1:4)
#距離計算;stats, amapパッケージを利用
#列
RowDist <- as.dendrogram(hclust(Dist(TestData, method = "pearson", nbproc = 200),
method = "complete"))
#行
ColDist <- as.dendrogram(hclust(Dist(t(TestData), method = "pearson", nbproc = 200),
method = "complete"))
##############################
##########デンドログラムの体裁,色,サイズを変更:setコマンド###########
#set(dendrogramオブジェクト, labels_xxxオプション, オブションに合わせた設定)
#setコマンド最下部にまとめてプロットコマンド
#内容の変更:labelsオプション
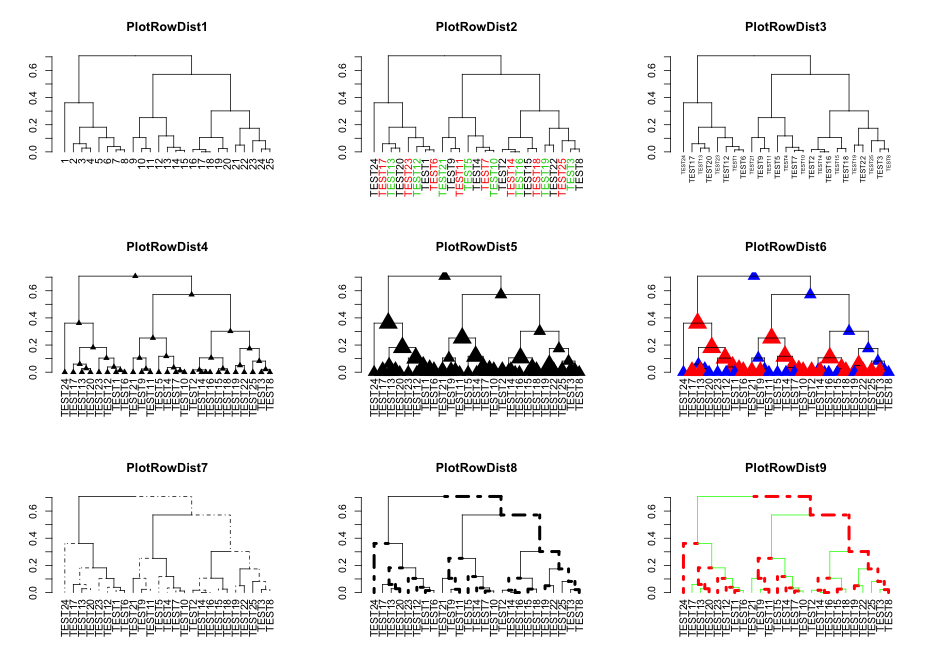
PlotRowDist1 <- set(RowDist, "labels", c(1:50))
#色の変更:labels_colオプション
PlotRowDist2 <- set(RowDist, "labels_col", c(1, 2, 3))
#文字サイズの変更:labels_cexオプション
PlotRowDist3 <- set(RowDist, "labels_cex", c(0.5, 0.7))
#ノードにドットを追加:nodes_pchオプション
#末端のみはleaves_pchオプション
PlotRowDist4 <- set(RowDist, "nodes_pch", 17)
###PlotRowDist5-6まで順番にオプションを追加###
#条件はc()で複数設定できます
#ドットのサイズを変更:nodes_cexオプション
#末端のみはleaves_cexオプション
PlotRowDist5 <- set(PlotRowDist4, "nodes_cex", c(2, 3))
#ドットの色を変更:nodes_colオプション
#末端のみはleaves_colオプション
PlotRowDist6 <- set(PlotRowDist5, "nodes_col", c("blue", "red"))
###############################################
###PlotRowDist7-9まで順番にオプションを追加###
#線の種類を設定:branches_ltyオプション
PlotRowDist7 <- set(RowDist, "branches_lty", c(4, 7))
#線の種類を設定:branches_lwdオプション
PlotRowDist8 <- set(PlotRowDist7, "branches_lwd", c(4, 1))
#線の色を設定:branches_colオプション
PlotRowDist9 <- set(PlotRowDist8, "branches_col", c("red", "green"))
###PlotRowDist1-9をプロット####################
par(mfrow = c(3,3))
#Mac日本語文字化け防止
#par(family = "HiraKakuProN-W3")
#プロット
for(n in 1:9){
plot(eval(parse(text = paste0("PlotRowDist", n))), main = paste0("PlotRowDist", n))
}
###############################################
#指定したクラスタに分類:branches_k_colorオプション
#valuオプションで色を指定できます
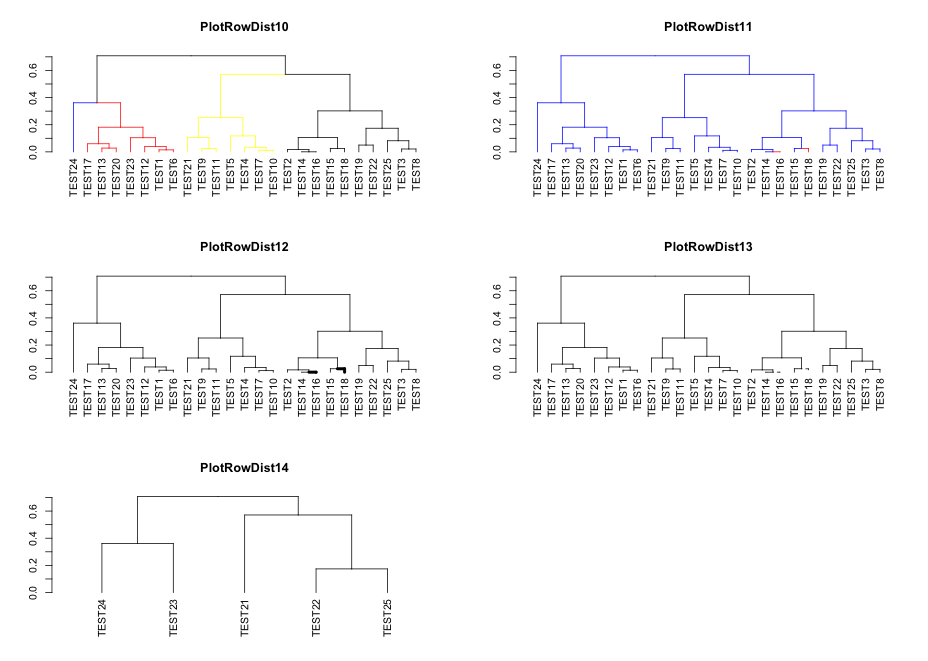
PlotRowDist10 <- set(RowDist, "branches_k_color",
value = c("blue", "red", "yellow", "black"), k = 4)
#指定したラベル内容のノードの色,太さ,線種変更:by_labels_branches_XXXオプション
#色の変更:by_labels_branches_col
PlotRowDist11 <- set(RowDist, "by_labels_branches_col",
value = c("TEST16", "TEST18"), TF_values = c("red", "blue"))
#線の太さを変更:by_labels_branches_col
PlotRowDist12 <- set(RowDist, "by_labels_branches_lwd",
value = c("TEST16", "TEST18"), TF_values = c(4, 1))
#線の太さを変更:by_labels_branches_lty
PlotRowDist13 <- set(RowDist, "by_labels_branches_lty",
value = c("TEST16", "TEST18"), TF_values = c(4, 1))
###その他の装飾コマンド#####
#指定したデータを取り除く:pruneコマンド
PlotRowDist14 <- prune(RowDist, leaves = paste0("TEST", 1:20))
###PlotRowDist10-14をプロット####################
par(mfrow = c(3, 2))
#Mac日本語文字化け防止
#par(family = "HiraKakuProN-W3")
#プロット
for(n in 0:4){
plot(eval(parse(text = paste0("PlotRowDist1", n))), main = paste0("PlotRowDist1", n))
}
###############################################
#プロット領域の初期化
par(mfrow = c(1, 1))
#指定したクラスタに分類し枠で囲む:rect.dendrogramコマンド
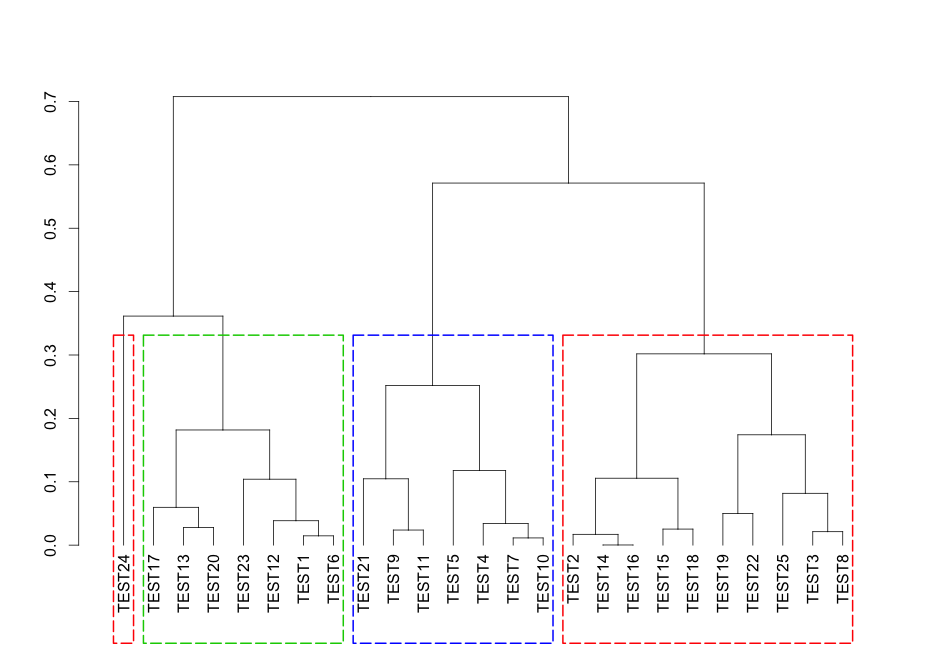
plot(RowDist)
PlotRowDist15 <- rect.dendrogram(RowDist, k = 4, border = c(2, 3, 4), lty = 5, lwd = 2)
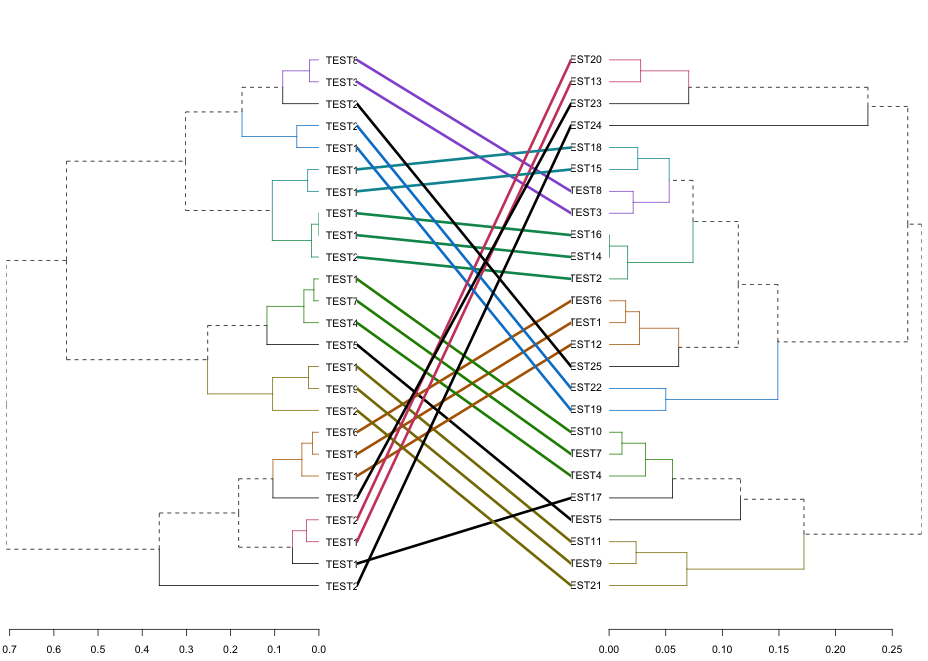
#デンドログラムを並べて表示の準備:dendlistコマンド
#結合方式を変えて比較
RowDist1 <- as.dendrogram(hclust(Dist(TestData, method = "pearson", nbproc = 200),
method = "complete"))
RowDist2 <- as.dendrogram(hclust(Dist(TestData, method = "pearson", nbproc = 200),
method = "average"))
RowDist1_2 <- dendlist(RowDist1, RowDist2)
#デンドログラムを並べて比較:tanglegramコマンド
tanglegram(RowDist1_2, common_subtrees_color_branches = TRUE)
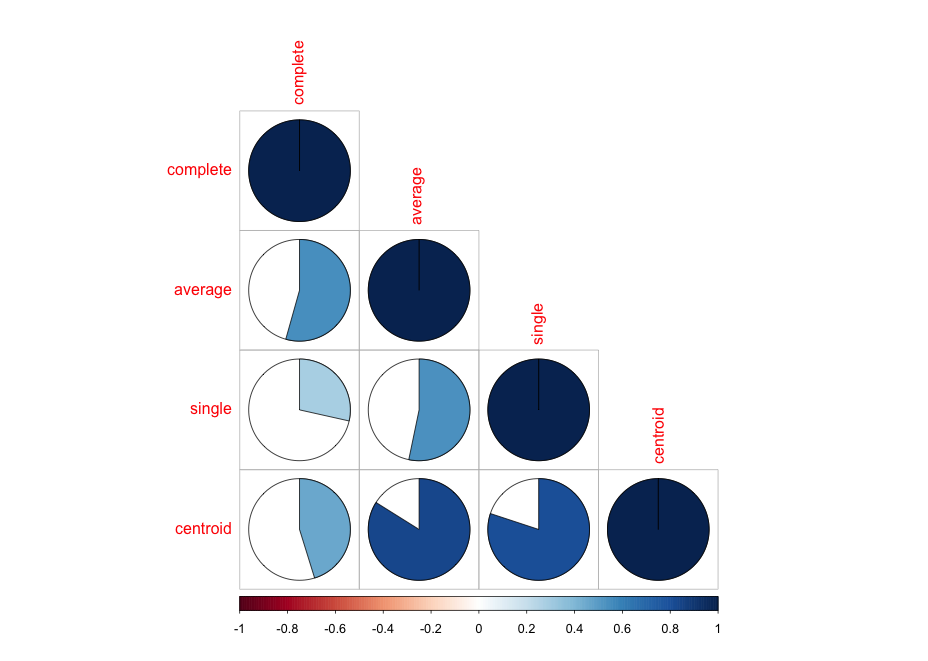
#corrplotパッケージを利用した結合方法の相関:cor.dendlistコマンド
#結合方式の準備
RowDist1 <- as.dendrogram(hclust(Dist(TestData, method = "pearson", nbproc = 200),
method = "complete"))
RowDist2 <- as.dendrogram(hclust(Dist(TestData, method = "pearson", nbproc = 200),
method = "average"))
RowDist3 <- as.dendrogram(hclust(Dist(TestData, method = "pearson", nbproc = 200),
method = "single"))
RowDist4 <- as.dendrogram(hclust(Dist(TestData, method = "pearson", nbproc = 200),
method = "centroid"))
RowDist1_2_3_4 <- dendlist("complete" = RowDist1, "average" = RowDist2, "single" = RowDist3, "centroid" = RowDist4)
#必要なパッケージのインストールと読み込み
#install.packages("corrplot")
library("corrplot")
#データのプロット
corrplot(cor.dendlist(RowDist1_2_3_4), "pie", "lower")
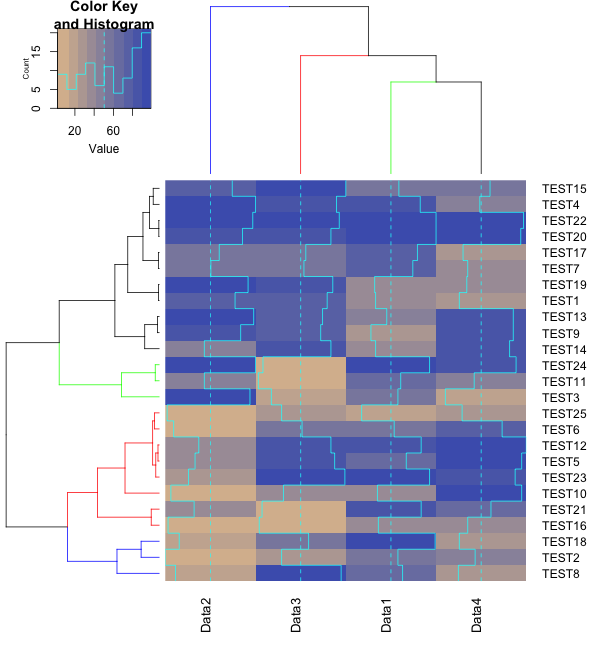
#########gplotsパッケージの「heatmap.2」コマンドとの組み合わせ#####
#install.packages("gplots")
if(!require("gplots", quietly = TRUE)){
install.packages("gplots");require("gplots")
}
#データ例の作成
#25*4のデータ
TestData <- matrix(sample(1:100, 100, replace = TRUE), 25, 4)
row.names(TestData) <- paste0("TEST", 1:25)
colnames(TestData) <- paste0("Data", 1:4)
#距離計算;stats, amapパッケージを利用
#列
RowDist <- as.dendrogram(hclust(Dist(TestData, method = "pearson", nbproc = 200),
method = "complete"))
#色を指定
RowDist <- set(RowDist, "branches_k_color",
value = c("blue", "red", "green", "black"), k = 4)
#行
ColDist <- as.dendrogram(hclust(Dist(t(TestData), method = "pearson", nbproc = 200),
method = "complete"))
#色を指定
ColDist <- set(ColDist, "branches_k_color",
value = c("blue", "red", "green", "black"), k = 4)
#heatmap.2コマンドでヒートマップをプロット
heatmap.2(TestData, Rowv = RowDist, Colv = ColDist, cexCol = 1,
col = colorRampPalette(c("#d9bb9c", "#4b61ba"))(10))出力
・PlotRowDist1-9をプロット
・PlotRowDist10-14をプロット
・rect.dendrogramコマンド
・tanglegramコマンド
・cor.dendlistコマンド
・gplotsパッケージの「heatmap.2」コマンドと組み合わせ
少しでも、あなたの解析が楽になりますように!!


