健康管理や研究目的でのスマートウォッチの利用を目にすることが多くなってきました。スマートウォッチは各社から販売されていますが、マラソンランナーなどに人気のGarmin(ガーミン)で得られるデータをRで読み込むのに便利な「FITfileR」パッケージを紹介します。
GarminはGarmin Connectと連携することで、スマートフォンのアプリやGarmin Connectのサイトで各種データを確認することが出来ます。
Garmin Connectのサイトで入手できるデータは「fitファイル」というバイナリファイルです。
Flexible and Interoperable Data Transfer (FIT) SDK:https://developer.garmin.com/fit/overview/
「fitファイル」のプロトコルが公開されていますので、興味ある方は「readBin」コマンドでファイルを解析してはいかがでしょうか。
なお、実行コマンドでも紹介していますが、「fitファイル」の時間起点は「December 31, 1989 UTC(631065600)」なので、「timestamp_16」は631065600をオフセットに使用することに注意です。
健康管理や研究目的でGarminのスマートウォッチの利用を検討してはいかがでしょうか。使用しているGarminnはVÍVOSMART 5です。睡眠やストレスなどの色々な健康情報を取得できます。
パッケージバージョンは0.1.5。実行コマンドはwindows 11のR version 4.2.1で確認しています。
fitファイルの入手
GarminとGarmin Connectの連携が終了している前提です。連携方法はGarminサイトなどで確認してください。
まずはGarmin Connectにアクセスします。
Garmin Connect:https://connect.garmin.com/signin/
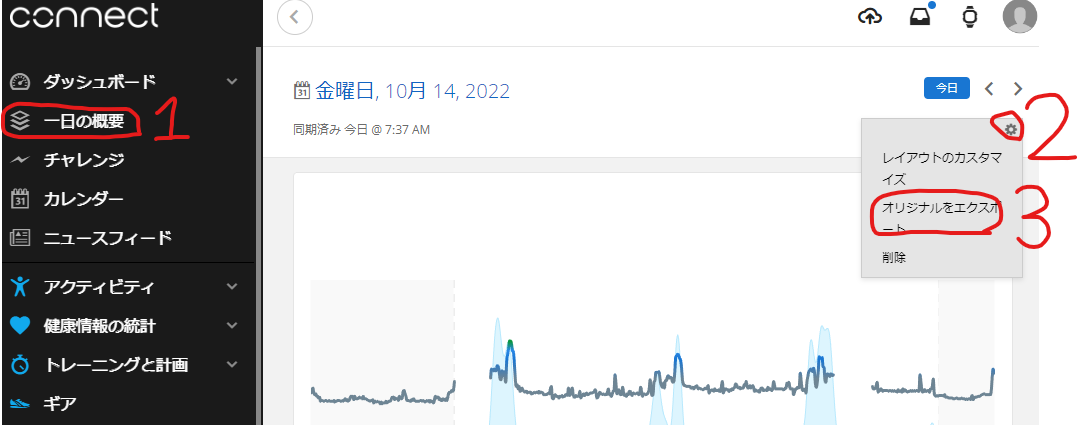
アクセス後「fitファイル」は以下の写真のステップで入手可能です。指定日ごとの「fitファイル」がzipで圧縮されたファイルを入手できます。
パッケージのインストール
下記コマンドを実行してください。
#パッケージのインストール
if(!requireNamespace("remotes")) {
install.packages("remotes")
}
remotes::install_github("grimbough/FITfileR")実行コマンドの紹介
詳細はコマンド、各パッケージのヘルプを確認してください。Garmin Connectから入手したzipファイルを展開して利用してください。展開後のフォルダに含まれる、全WELLNESS.fitを対象に処理して、心拍数、ストレスレベルのデータを入手するコマンド例です。対象「fitファイル」を「listMessageTypes」コマンドでメッセージを確認し、「getMessagesByType」コマンドで目的のデータを入手することが可能です。
#パッケージの読み込み
library("FITfileR")
#tidyverseパッケージがなければインストール
if(!require("tidyverse", quietly = TRUE)){
install.packages("tidyverse");require("tidyverse")
}
#lubridateパッケージがなければインストール
#https://www.karada-good.net/analyticsr/r-467/
if(!require("lubridate", quietly = TRUE)){
install.packages("lubridate");require("lubridate")
}
#wellnessファイルの読み込み
#ReadFit <- readFitFile(file.choose())
#データの確認
#listMessageTypes(ReadFit)
#[1] "file_id" "event" "device_info" "software"
#[5] "monitoring" "monitoring_info" "ohr_settings" "stress_level"
###連続処理するための準備#####
#展開したfitファイルのフォルダを作業ディレクトリにする
setwd(choose.dir())
#ファイル名を取得
GetFileNames <- list.files()
#_WELLNESS.fitを対象にする
AnaFileNames <- str_subset(GetFileNames, "WELLNESS")
#データ格納用引数
ActivityData <- NULL
HartRateData <- NULL
StressData <- NULL
###繰り返し処理#####
for(i in seq(AnaFileNames)){
ReadFit <- readFitFile(AnaFileNames[[i]])
if("event" %in% listMessageTypes(ReadFit)){
###活動内容,心拍数の取得#####
MonitoringData <- getMessagesByType(ReadFit,
message_type = "monitoring")
#日付けの一括変換
for(n in seq(MonitoringData)){
#日付けデータの変換
MonitoringData[[n]] <-
MonitoringData[[n]] %>%
mutate_if(is.POSIXt, ~with_tz(., tz = "Asia/Tokyo"))
}
###timestamp_16の変換#####
#参考資料:https://developer.garmin.com/fit/cookbook/datetime/
#fitファイルの時間起点は「December 31, 1989 UTC(631065600)」なので、
#「timestamp_16」は631065600をオフセットに使用する
#fitファイル開始時間の取得
TimCre <- as.numeric(as.POSIXct(file_id(ReadFit)[[2]])[[1]]) - 631065600
#変換処理
for(n in seq(MonitoringData)){
#日付けデータの変換
MonitoringData[[n]] <-
MonitoringData[[n]] %>%
mutate_at(vars(matches("timestamp_16")),
function(.){
TimCre +
#bbase::it演算:pythonで「X & 0xffff」
bitwAnd((. - TimCre), 0xffff) + 631065600 %>%
lubridate::as_datetime() %>%
lubridate::with_tz(tz = "Asia/Tokyo")})
}
########
###アクティビティデータを取得#####
#"current_activity_type_intensity"を含む列名のlist番号を取得
ActivityNo <- which(sapply(MonitoringData,
function(x) "current_activity_type_intensity" %in% colnames(x)))
for(For_ActiNo in seq(length(ActivityNo))){
if("timestamp" %in% colnames(MonitoringData[[ActivityNo[For_ActiNo]]])){
ActivityData <- bind_rows(ActivityData,
MonitoringData[[ActivityNo[For_ActiNo]]])
}else{
MonitoringData[[ActivityNo[For_ActiNo]]] %>%
select("timestamp_16", "current_activity_type_intensity") %>%
bind_rows(ActivityData)
}}
###心拍数データを取得#####
#"heart_rate"を含む列名のlist番号を取得
HartRateNo <- which(sapply(MonitoringData, function(x) "heart_rate" %in% colnames(x)))
#HartRate <- rbind(HartRate, MonitoringData[[HartRateNo]])
HartRateData <- bind_rows(HartRateData, MonitoringData[[HartRateNo]])
###ストレスレベルの取得#####
StressLevelData <- getMessagesByType(ReadFit,
message_type = "stress_level") %>%
mutate_if(is.POSIXt, ~with_tz(., tz = "Asia/Tokyo"))
#StressData <- rbind(StressData, StressLevelData)
StressData <- bind_rows(StressData, StressLevelData)
}
}
#列名を整える
colnames(ActivityData) <- c("Time_Stamp", "Activity_type_intensity")
colnames(HartRateData) <- c("Time_Stamp", "Hart_Rate")
colnames(StressData) <- c("Time_Stamp", "Stress_Level")
#オブジェクト:"ActivityData","HartRate","StressData"以外を削除
remove(list = ls()[!ls() %in% c("ActivityData", "HartRateData", "StressData")])
########出力例
・心拍データをプロットする
#tidyverseパッケージがなければインストール
if(!require("tidyverse", quietly = TRUE)){
install.packages("tidyverse");require("tidyverse")
}
ggplot(HartRateData, aes(x = Time_Stamp, y = Hart_Rate)) +
geom_area(fill = "#4b61ba") +
labs(title = "Hart_Rate") +
theme_minimal()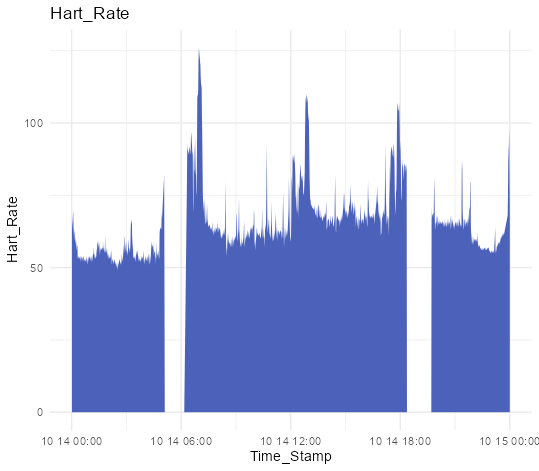
・ストレスレデータをプロットする
#tidyverseパッケージがなければインストール
if(!require("tidyverse", quietly = TRUE)){
install.packages("tidyverse");require("tidyverse")
}
ggplot(StressData, aes(x = Time_Stamp, y = Stress_Level)) +
geom_area(fill = "#a87963") +
labs(title = "Stress_Level") +
theme_minimal()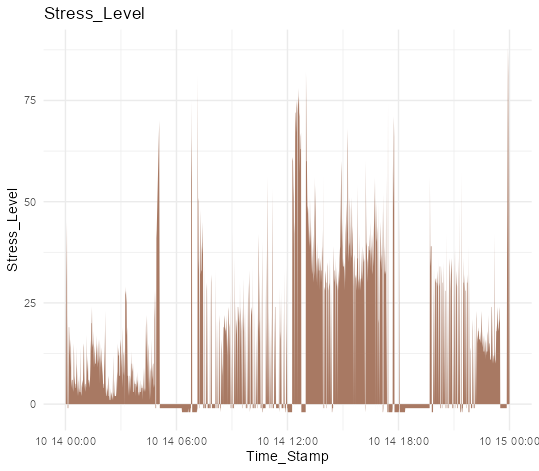
少しでも、あなたの解析が楽になりますように!!