とにかく、データセットが必要な方にオススメのパッケージです。100個のデータセットが収録されています。100個もあれば目的にあったデータセットが見つかるかもしれません。
すべて紹介するのは確認も大変なので、ヘルプの頭から50個のデータセットをstrコマンドで出力した結果を紹介します。データセットの詳細はパッケージヘルプを確認してください。
バージョンは1.0。実行コマンドはR version 3.2.2で確認しています。
パッケージのインストール
下記コマンドを実行してください。
[code language=”R”]
#パッケージのインストール
install.packages(“gpk”)
[/code]
実行コマンド
詳細はコメント、パッケージヘルプを確認してください。
[code language=”R”]
#パッケージの読み込み
library(“gpk”)
#No1.AIDSデータセット
data(AIDS)
str(AIDS)
‘data.frame’: 72 obs. of 5 variables:
$ SR.NO : int 1 2 3 4 5 6 7 8 9 10 …
$ PRE.TEST : int 12 13 8 9 10 7 10 12 12 3 …
$ POST.TEST: int 19 19 19 19 18 19 18 19 19 14 …
$ Sub.Code : int 1 1 1 1 1 1 1 1 1 1 …
$ Subject : Factor w/ 5 levels “Bot “,”Chem “,..: 2 2 2 2 2 2 2 2 2 2 …
#No2.AirPollutionデータセット
data(AirPollution)
str(AirPollution)
‘data.frame’: 151 obs. of 11 variables:
$ PM10 : num 120.7 140.2 107 88.8 75 …
$ Pb : num 2.67 3.49 1.31 0.76 0.6 0.52 1.17 2.53 0.95 0.78 …
$ Cd : num 0.01 0.02 0.01 0 0 0 0 0 0 0 …
$ Cu : num 0.53 0.1 0.19 0.33 0.41 0.69 0.13 0.09 0.07 0.07 …
$ Cr : num 0.61 0.91 0.48 0.39 1.54 0.42 0.24 0.38 0.19 0.2 …
$ Zn : int 0 0 0 0 0 0 0 0 0 0 …
$ NOx : num 84.2 88.8 81.5 61.8 48.7 …
$ SO2 : num 40.4 39.5 48.3 27.4 19.2 …
$ Site : Factor w/ 3 levels “Bhosari”,”Mandai”,..: 2 2 2 2 2 2 2 2 2 2 …
$ Date : Date, format: “2013-02-07” “2013-02-14” …
$ Season: Factor w/ 3 levels “Monsoon “,”Summer “,..: 2 2 2 2 2 2 2 2 2 2 …
#No3.AizawlCancerデータセット
data(AizawlCancer)
str(AizawlCancer)
‘data.frame’: 19 obs. of 3 variables:
$ Site : Factor w/ 19 levels “Breast”,”Colon”,..: 15 8 10 1 13 5 2 12 7 3 …
$ Female: int 244 130 29 119 42 12 32 23 21 23 …
$ Male : int 569 101 117 2 35 46 29 33 26 9 …
#No4.Allergyデータセット
data(Allergy)
str(Allergy)
‘data.frame’: 7 obs. of 4 variables:
$ ProdA: int 42 59 87 44 93 11 53
$ ProdB: int 72 50 24 59 83 28 27
$ ProdC: int 97 49 86 84 88 29 103
$ ProdD: int 23 42 18 23 24 68 98
#No5.Asthma1データセット
data(Asthma1)
str(Asthma1)
‘data.frame’: 12 obs. of 4 variables:
$ Log_Concentration_Histamine : num 7.3 7.2 6.9 6.6 7.3 …
$ Response_Without_Curcuma_Longa: int 12 17 30 32 10 15 35 48 8 10 …
$ Response_With_Curcuma_Longa : int 1 1 4 15 1 2 4 17 1 2 …
$ Group : Factor w/ 3 levels “Set 1 “,”Set2 “,..: 1
#No6.Asthma2データセット
data(Asthma2)
str(Asthma2)
‘data.frame’: 10 obs. of 4 variables:
$ Animal.code: Factor w/ 10 levels “I”,”II”,”III”,..: 1 2 3 4 6 7 8 9 5 10
$ Before : int 175 150 125 175 125 200 175 175 125 150
$ After : int 275 250 200 250 200 225 225 200 175 200
$ Group : Factor w/ 2 levels “Control “,”Treatment “: 1 1 1 1 1 2 2 2 2 2
#No7.Asthma3データセット
data(Asthma3)
str(Asthma3)
‘data.frame’: 15 obs. of 3 variables:
$ Treatment : Factor w/ 3 levels “Control”,”Curcuma longa”,..: 1 1 1 1 1 3 3 3 3 3 …
$ Animal.Code: int 1 2 3 4 5 6 7 8 9 10 …
$ Response : int 77 83 76 78 80 24 27 25 26 24 …
#No8.Asthma4データセット
data(Asthma4)
str(Asthma4)
‘data.frame’: 42 obs. of 4 variables:
$ Extent_of_Exposure: int 0 0 0 0 0 0 0 25 25 25 …
$ Years_Exposure : Factor w/ 7 levels “0-7″,”12 to 15”,..: 1 7 2 3 4 5 6 1 7 2 …
$ Death_Count : int 10 12 19 31 35 48 73 17 17 17 …
$ At_Risk_Count : int 262 243 240 237 233 227 220 313 290 285 …
#No9.atombombデータセット
data(atombomb)
str(atombomb)
‘data.frame’: 42 obs. of 4 variables:
$ Extent_of_Exposure: int 0 0 0 0 0 0 0 25 25 25 …
$ Years_Exposure : Factor w/ 7 levels “0-7″,”12 to 15”,..: 1 7 2 3 4 5 6 1 7 2 …
$ Death_Count : int 10 12 19 31 35 48 73 17 17 17 …
$ At_Risk_Count : int 262 243 240 237 233 227 220 313 290 285 …
#No10.Bacteriaデータセット
data(Bacteria)
str(Bacteria)
‘data.frame’: 300 obs. of 5 variables:
$ Response: num -5.55 -5.15 -5.05 -5.22 -5.17 -4.81 -5.46 -1.58 -4.94 -5.12 …
$ Salt : Factor w/ 5 levels “0%”,”1%”,”2%”,..: 1 2 1 3 2 4 1 3 5 2 …
$ Lipid : Factor w/ 5 levels “0%”,”10%”,”15%”,..: 1 1 5 1 5 1 2 5 1 2 …
$ pH : int 3 3 3 3 3 3 3 3 3 3 …
$ Temp : int 0 0 0 0 0 0 0 0 0 0 …
#No11.BambooGrowthデータセット
data(BambooGrowth)
str(BambooGrowth)
‘data.frame’: 595 obs. of 5 variables:
$ Compartment : int 6 6 6 6 6 6 6 6 6 6 …
$ Locality_Block : int 5 5 5 5 5 5 5 5 5 5 …
$ Transect_Number: int 1 1 1 1 1 1 1 1 1 1 …
$ Old_Shoots : int 7 4 5 4 9 12 4 5 3 3 …
$ New_Shoots : int 0 0 0 0 0 0 0 0 0 0 …
#No12.Bamboolifeデータセット
data(Bamboolife)
str(Bamboolife)
‘data.frame’: 16 obs. of 2 variables:
$ Age : int 1 2 3 4 5 6 7 8 9 10 …
$ Survivors: int 439 438 433 427 410 380 340 286 229 168 …
#No13.Bananabatsデータセット
data(Bananabats)
str(Bananabats)
‘data.frame’: 16 obs. of 5 variables:
$ Date : Date, format: “0093-08-28” “1993-09-09” …
$ Period : int 1 1 1 1 1 2 2 2 2 2 …
$ Number.caught : int 40 44 49 27 36 24 34 26 24 23 …
$ Number.observed: int 40 46 50 34 38 30 35 29 25 24 …
$ KTBA : int 84 75 66 60 58 66 58 52 50 50 …
#No14.BANKデータセット
data(BANK)
str(BANK)
‘data.frame’: 245 obs. of 20 variables:
$ Serial_Number : int 1 2 3 4 5 6 7 8 9 10 …
$ Response : int 0 0 0 0 0 0 0 0 0 0 …
$ Branch : Factor w/ 10 levels “B1″,”B10″,”B2”,..: 1 1 1 1 1 1 1 1 1 1 …
$ Occupation : int 6 1 0 6 1 2 0 4 0 3 …
$ Age : int 5 5 5 4 2 3 5 1 5 4 …
$ Sex : int 2 1 1 2 1 1 2 1 1 1 …
$ Pleasant_Ambiance : int 9 10 4 4 10 7 7 7 6 7 …
$ Comfortable_seating_arrangement : int 5 8 8 6 8 8 7 7 5 6 …
$ Immediate_attenttion : int 8 8 7 6 6 6 5 6 7 5 …
$ Good_Response_on_Phone : int 6 6 8 8 10 7 7 7 7 8 …
$ Errors_in_Passbook_entries : int 8 10 5 6 8 8 5 7 7 9 …
$ Time_to_issue_cheque_book : int 0 8 7 4 8 7 8 9 9 7 …
$ Time_to_sanction_loan : int 6 9 4 4 5 7 5 5 5 8 …
$ Time_to_clear_outstation_cheques: int 4 6 5 5 7 7 5 4 7 4 …
$ Issue_of_clean_currency_notes : int 6 8 7 6 8 6 5 6 7 5 …
$ Facility_to_pay_bills : int 3 6 6 6 7 7 7 8 7 8 …
$ Distance_to_residence : int 10 8 8 9 10 8 7 9 9 7 …
$ Distance_to_workplace : int 9 7 7 6 10 7 6 7 8 9 …
$ Courteous_staff_behaviour : int 8 10 9 9 5 8 5 8 8 9 …
$ Enough_parking_place : int -2 10 6 5 8 7 5 7 8 9 …
#No15.Barleyheightデータセット
data(Barleyheight)
str(Barleyheight)
‘data.frame’: 9 obs. of 23 variables:
$ Years : int 1974 1975 1976 1977 1978 1979 1980 1981 1982
$ Genotype1 : num 81 67.3 71.5 64.3 55.8 84.9 86.2 88 72
$ Genotype2 : num 72.3 60.3 60.8 55.3 48.8 78.1 80.4 85.3 69.8
$ Genotype3 : num 79.3 67.8 64.8 57.5 46.8 80.2 81.8 87.8 71.8
$ Genotype4 : num 88.5 70.8 76.3 69.5 64 90.8 97.3 97.8 86
$ Genotype5 : num 78.5 67.5 72.5 61 50.3 78.7 82.7 87.3 66
$ Genotype6 : num 89.3 74.5 80.5 67.8 60.8 86.3 90.2 100 81.3
$ Genotype7 : num 94.3 73 80.3 68.5 63.8 …
$ Genotype8 : num 88.8 63.8 66.8 78.5 70.3 …
$ Genotype9 : num 91.3 67 73.8 75.8 71.5 …
$ Genotype10 : num 91.8 65.5 77 80 73.5 …
$ Genotype11 : num 86 69.8 73.8 77.3 75.5 …
$ Genotype12 : num 91 71.8 81 65.5 54.5 87.9 84.8 91.8 77.8
$ Genotype13 : num 75.5 56.5 67 64.3 58.8 86.7 85.2 91.8 76
$ Genotype14 : num 96.8 81.5 86.3 73.3 59.3 97 96.1 95.8 90.3
$ Genotype15 : num 97 83.3 86.8 72 49.3 91.3 94.6 95.5 80.8
$ Sowing.day.Number.days.since.April1: int 17 21 26 48 35 39 35 38 27
$ Rainfall1 : num 0 1.66 0.8 0.21 0.37 1.35 0.67 0.23 2.5
$ Rainfall2 : num 0.16 1.84 2.05 3.62 0.66 3.92 3.45 3.2 3.29
$ Rainfall3 : num 1.59 1.68 0.81 0.36 0.75 1.05 2.75 4.36 0.01
$ Rainfall4 : num 2.65 0.08 0.4 3.46 6.72 2.22 6.2 3.01 2.76
$ Rainfall5 : num 1.7 0.02 1.02 0.15 3.32 0.86 1.05 4.14 3.31
$ Rainfall6 : num 2.21 0.01 1.06 4.65 0.88 2.49 3.6 0.54 2.54
#No16.BatGroupデータセット
data(BatGroup)
str(BatGroup)
‘data.frame’: 6 obs. of 9 variables:
$ Month : Factor w/ 6 levels “April”,”August”,..: 2 6 5 1 4 3
$ GS_1 : int 15 33 17 17 19 30
$ GS_2 : int 4 10 2 8 9 9
$ GS_3 : int 1 3 4 2 1 9
$ GS_4 : int 4 2 2 3 3 2
$ GS_5 : int 2 2 1 1 1 1
$ GS_6 : int 1 3 0 1 1 0
$ GS_7 : int 1 1 2 0 0 0
$ GS_GT_7: int 0 1 1 0 0 0
#No17.Batrecaptureデータセット
data(Batrecapture)
str(Batrecapture)
‘data.frame’: 11 obs. of 2 variables:
$ Number.recapture : int 1 2 3 4 5 6 7 8 9 10 …
$ Number.individuals: int 5061 2163 949 417 171 81 44 15 4 1 …
#No18.Biodegradationデータセット
data(Biodegradation)
str(Biodegradation)
‘data.frame’: 16 obs. of 5 variables:
$ pH : int 5 5 5 5 5 5 5 5 7 7 …
$ Temp : int 30 30 30 30 40 40 40 40 30 30 …
$ Inoculum : Factor w/ 4 levels “H”,” H”,”L”,..: 4 4 2 2 4 4 2 2 4 4 …
$ Aeration : Factor w/ 2 levels “No”,”Yes”: 1 2 1 2 1 2 1 2 1 2 …
$ Percent.Removal: num 13.7 24.9 60.9 78.6 15.3 …
#No19.birdextinctデータセット
data(birdextinct)
str(birdextinct)
‘data.frame’: 18 obs. of 4 variables:
$ Site : int 1 2 3 4 5 6 7 8 9 10 …
$ Area : num 185.8 105.8 30.7 8.5 4.8 …
$ Species_at_risk : int 75 67 66 51 28 20 43 31 28 32 …
$ Number_of_Species_extinct: int 5 3 10 6 3 4 8 3 5 6 …
#No20.BirthDeathデータセット
data(BirthDeath)
str(BirthDeath)
‘data.frame’: 27 obs. of 3 variables:
$ Year : Factor w/ 27 levels “1901-1910″,”1911-1920”,..: 1 2 3 4 5 6 7 8 9 10 …
$ Birth.rate: num 49.2 48.1 46.6 45.2 39.9 41.7 41.2 36.9 36.6 34.6 …
$ death.rate: num 42.6 47.2 36.3 31.2 27.4 22.8 19 14.9 16.9 15.5 …
#No21.BPSYSデータセット
data(BPSYS)
str(BPSYS)
‘data.frame’: 35 obs. of 8 variables:
$ Pat_no : int 5 8 35 33 29 13 1 7 31 25 …
$ Age : int 64 46 60 65 52 NA 34 48 53 61 …
$ Sex : Factor w/ 2 levels “F”,”M”: 1 1 2 2 1 2 1 2 1 2 …
$ Duration_of_hypertension_yrs: num 0.33 0.83 0.42 1 1 2 2 6 0.5 0.58 …
$ Duration_of_diabetes_yrs : num 15 15 10 8 1 2 2 14 2 4 …
$ BaselineSystolic_BP : int 150 140 140 150 160 150 168 140 140 160 …
$ Week_8_Systolic_BP : int 130 140 120 130 140 120 140 120 130 150 …
$ Drug : Factor w/ 2 levels “Al”,”Ay”: 2 2 2 2 2 2 2 2 2 2 …
#No22.Butterfliesデータセット
data(Butterflies)
str(Butterflies)
‘data.frame’: 44 obs. of 9 variables:
$ Serial_Number : int 1 2 3 4 5 6 7 8 9 10 …
$ Area : Factor w/ 8 levels “Central Himalaya”,..: 2 8 8 8 8 8 8 8 1 3 …
$ Locality : Factor w/ 44 levels “Andaman Nikobar”,..: 11 44 13 36 9 25 26 20 4 31 …
$ Total_Species_count: int 1439 417 228 299 148 323 146 371 623 962 …
$ Skippers : int 307 63 25 41 22 54 14 52 125 211 …
$ Swallow_tails : int 94 31 23 21 11 23 10 26 43 69 …
$ Whites_Yellows : int 99 42 37 34 19 32 13 37 49 57 …
$ Blues : int 458 129 56 88 42 88 44 109 185 284 …
$ Brush_Footed : int 482 152 87 115 54 126 65 147 221 342 …
#No23.Chitalparasiteデータセット
data(Chitalparasite)
str(Chitalparasite)
‘data.frame’: 66 obs. of 6 variables:
$ Sarcocystis_Indicator: int 0 1 0 0 0 0 1 0 0 0 …
$ Sanctuary_Indicator : int 2 2 2 2 2 2 2 2 2 2 …
$ Predator_Indicator : int 2 2 2 2 2 2 2 2 2 2 …
$ Tissue_Indicator : int 2 1 2 2 2 2 1 1 2 2 …
$ SEX : Factor w/ 2 levels “F”,”M”: 2 2 1 1 2 1 1 1 1 2 …
$ YEAR : int 2001 2001 2001 2001 2001 2001 2001 2001 2001 2001 …
#No24.cloudseedデータセット
data(cloudseed)
str(cloudseed)
‘data.frame’: 52 obs. of 2 variables:
$ Rainfall : num 1203 830 372 346 321 …
$ Seeded.Indicator: int 0 0 0 0 0 0 0 0 0 0 …
#No25.Cosmetic1データセット
data(Cosmetic1)
str(Cosmetic1)
‘data.frame’: 48 obs. of 3 variables:
$ Treatment: Factor w/ 3 levels “A”,”B”,”C”: 2 1 2 2 2 1 1 1 2 2 …
$ Initial : int 188 164 191 212 156 154 219 162 243 171 …
$ Change : num 32.5 262.5 49.5 412.5 214.5 …
#No26.COWSDATAデータセット
data(COWSDATA)
str(COWSDATA)
‘data.frame’: 10 obs. of 7 variables:
$ Time : Factor w/ 10 levels “0–6″,”12–18”,..: 1 10 2 3 4 5 6 7 8 9
$ Sillod_Insemination_C1: int 119 2145 1079 189 1779 733 258 114 94 44
$ Sillod_Conception_C1 : int 11 235 117 17 110 54 15 5 1 2
$ Sillod_Insemination_C2: int 22 999 1088 93 1011 755 320 26 107 27
$ Sillod_Conception_C2 : int 0 214 114 7 73 87 34 3 10 2
$ Sillod_Insemination_C3: int 843 1369 741 286 1668 859 423 99 210 82
$ Sillod_Conception_C3 : int 177 220 145 62 240 123 40 8 26 18
#No27.Crackデータセット
data(Crack)
str(Crack)
‘data.frame’: 17 obs. of 4 variables:
$ Right_Heel_Change_Grade : num -0.33 -1.33 -3.06 -1.92 -2 -1.29 -1.22 -1.56 -1 -1.25 …
$ Right_Heel_Change_Length: num -0.32 -0.61 -1.04 -0.31 -0.27 -0.39 -0.18 -0.93 -0.13 -0.21 …
$ Left_Heel_Change_Grade : num -0.67 -0.95 -2.64 -0.91 -1.25 -1.33 -0.73 -1.63 -1.5 -1 …
$ Leftt_Heel_Change_Length: num -0.36 -0.42 -0.78 -0.24 -0.3 -0.58 -1.01 -0.63 -0.5 -0.44 …
#No28.Crimeデータセット
data(Crime)
str(Crime)
‘data.frame’: 18 obs. of 2 variables:
$ Delinquency.index : num 26.2 33 17.5 25.2 20.3 …
$ Intelligence.Quotient: int 110 89 102 98 110 98 122 119 120 92 …
#No29.DroughtStressデータセット
data(DroughtStress)
str(DroughtStress)
‘data.frame’: 33 obs. of 25 variables:
$ Variety : Factor w/ 11 levels “296B”,”E36-1″,..: 2 2 2 3 3 3 6 6 6 4 …
$ RWC_00 : num 96.8 96.2 95.8 94.8 96.1 92.9 93.8 95 97.6 99.2 …
$ CO2FIx_00: num 2.72 2.92 3.4 8.03 7.09 6.08 5.84 6.02 5.21 1.55 …
$ Cond_00 : num 59.5 89.8 71 73.2 78.3 …
$ IntCO2_00: num 337 341 355 194 242 …
$ RWC_05 : num 97.4 97.8 97.4 92.7 98.2 96.8 96.7 94.8 94.2 98.9 …
$ CO2FIx_05: num 6.13 7.52 7.06 5.61 7.56 6.39 8.78 6.73 6.24 8.85 …
$ Cond_05 : num 79.3 74.6 63.6 58.9 85.8 69.9 78.4 73.5 77.9 123 …
$ IntCO2_05: num 281 225 260 193 190 …
$ RWC_10 : num 95.7 94.2 94.2 98.9 95.5 97 91.3 86.4 92.5 97.4 …
$ CO2FIx_10: num 4.61 5.3 4.32 7.84 8.77 8.06 3.51 7.67 7.99 7.5 …
$ Cond_10 : num 48.9 54.7 34.7 62.5 72 75.8 37.1 62.8 72.4 93.6 …
$ IntCO2_10: num 249 218 226 153 155 …
$ RWC_15 : num 89.1 90.6 91.2 95.4 96.9 91.8 88.6 92.1 94.4 93.8 …
$ CO2FIx_15: num 5.83 7.16 6.23 7.97 6.93 4.59 7.32 8.43 7.05 7.62 …
$ Cond_15 : num 51.2 83.3 55.6 86.1 82.3 68.1 54.4 73 64.9 55 …
$ IntCO2_15: num 185 214 252 213 235 …
$ RWC_20 : num 85.7 94.3 64.6 95.4 95 92.2 94.1 86.7 96.8 94.4 …
$ CO2FIx_20: num 3.73 5.85 3.19 5.64 5.96 5.56 5.17 7.36 7.63 4.44 …
$ Cond_20 : num 27 48.3 32.1 60.3 64.1 45.6 42.9 65 53.5 56.6 …
$ IntCO2_20: num 152 202 224 204 287 …
$ RWC_25 : num 87 65.6 70.7 46.9 94.9 64.8 78.9 63.3 86.8 87 …
$ CO2FIx_25: num 1.89 3.21 6.47 5.82 2.27 1.4 3.12 2.47 3.89 5.59 …
$ Cond_25 : num 16.6 42.5 50.5 36.1 28.4 21 31.1 36.8 26.4 50 …
$ IntCO2_25: num 292 272 194 183 262 …
#No30.Dunglifeデータセット
data(Dunglife)
str(Dunglife)
‘data.frame’: 55 obs. of 1 variable:
$ Decay: int 3 5 26 25 24 22 22 22 30 20 …
#No31.Earthquakeデータセット
data(Earthquake)
str(Earthquake)
‘data.frame’: 121 obs. of 10 variables:
$ Date : Date, format: “2001-01-26” “2001-01-26” …
$ Hours : int 14 15 18 19 23 3 3 4 9 10 …
$ Minutes : int 55 11 20 10 14 34 50 36 10 18 …
$ Magnitude_IMD : num 4 4.6 4.5 4 4 4 4.7 5.2 4.1 4.1 …
$ Magnitued_USGS : num NA 4.8 4.7 4.2 NA 4.1 4.7 4.8 NA NA …
$ Magnitude_NGRI : num 4 4.6 4.3 3.9 3.7 4.1 4.5 4.4 3.9 3.9 …
$ Coda_duration_1_mm : int 670 806 913 645 614 654 978 1079 576 683 …
$ Coda_duration_2_mm : int 631 746 775 528 539 535 764 849 495 613 …
$ Coda_duration_6_mm : int 284 405 371 318 396 279 452 654 270 296 …
$ Coda_duration_10_mm: int 245 338 335 288 237 231 357 527 196 258 …
#No32.Earthwormbiomassデータセット
data(Earthwormbiomass)
str(Earthwormbiomass)
‘data.frame’: 12 obs. of 5 variables:
$ Density: int 210 251 75 17 552 556 0 0 204 226 …
$ Biomass: num 15.1 22.2 6.1 2.3 83.2 60.4 0 0 26.3 31.1 …
$ Crop : Factor w/ 3 levels “Maize”,”Paddy and Pulses”,..: 1 1 1 1 2 2 2 2 3 3 …
$ Year : int 1998 1999 1998 1999 1998 1999 1998 1999 1998 1999 …
$ Soil : Factor w/ 2 levels “0-10 “,”10-20 “: 1 1 2 2 1 1 2 2 1 1 …
#No33.EarthwormSeasonデータセット
data(EarthwormSeason)
str(EarthwormSeason)
‘data.frame’: 46 obs. of 3 variables:
$ Month : Factor w/ 24 levels “01/00″,”01/99”,..: 9 9 11 11 13 13 15 15 17 19 …
$ Density: num 6 11 15 13.5 11 14.5 17.5 13.5 14 12 …
$ Biomass: num 2 4.5 6.5 9 8 11 17 14 12.5 11 …
#No34.elephantデータセット
data(elephant)
str(elephant)
‘data.frame’: 41 obs. of 2 variables:
$ Age_in_Years : int 27 28 28 28 28 29 29 29 29 29 …
$ Number_of_Matings: int 0 1 1 1 3 0 0 0 2 2 …
#No35.Euphorbiaceaeデータセット
data(Euphorbiaceae)
str(Euphorbiaceae)
‘data.frame’: 106 obs. of 4 variables:
$ Family : Factor w/ 1 level “EUPHORBIACEAE”: 1 1 1 1 1 1 1 1 1 1 …
$ Species_Name: Factor w/ 6 levels “Aporusa lindleyana”,..: 1 1 1 1 1 1 1 1 1 1 …
$ GBH : int 29 42 49 34 31 28 26 41 30 68 …
$ Height : int 10 20 20 15 10 10 15 10 15 30 …
#No36.Extruderデータセット
data(Extruder)
str(Extruder)
‘data.frame’: 49 obs. of 4 variables:
$ WEIGHT : num 4.46 4.46 4.45 4.43 4.41 4.43 4.45 4.51 4.4 4.38 …
$ EXTRUDER_RPM : int 60 59 60 60 60 60 59 59 59 59 …
$ CURRENT : int 69 70 70 72 68 69 70 71 67 70 …
$ Conveyer_Speed: int 70 69 69 70 70 69 69 69 69 69 …
#No37.Fairnessデータセット
data(Fairness)
str(Fairness)
‘data.frame’: 25 obs. of 3 variables:
$ Prod_A: num -0.25 -0.5 0 -0.75 -0.5 -0.5 -0.25 -0.5 -0.5 0 …
$ Prod_B: num -0.25 0.25 -0.5 -0.25 0 -0.25 -0.25 0 -0.5 -0.25 …
$ Prod_C: num -0.25 -0.5 -0.75 -0.75 -0.5 -0.25 0 -0.25 -0.25 -0.5 …
#No38.FAMILYデータセット
data(FAMILY)
str(FAMILY)
‘data.frame’: 288 obs. of 17 variables:
$ Serial_Number: int 1 2 3 4 5 6 7 8 9 10 …
$ Family_Code : int 11 12 13 14 15 16 17 18 19 110 …
$ FHT : num 167 166 169 163 165 167 163 167 164 166 …
$ MHT : num 161 164 166 158 160 164 157 162 160 164 …
$ Children : int 3 2 2 1 2 2 2 3 3 3 …
$ SEX_C1 : int 0 0 0 1 0 0 1 0 1 0 …
$ AGE_C1 : num 18 10 8 2 12 10 12 18 17 12 …
$ HT_C1 : num 156 115 110 59 124 117 122 158 158 127 …
$ SEX_C2 : int 1 0 1 NA 1 1 1 0 1 1 …
$ AGE_C2 : int 15 6 6 NA 7 6 9 15 14 10 …
$ HT_C2 : num 158 109 105 NA 108 103 114 154 152 129 …
$ SEX_C3 : int 0 NA NA NA NA NA NA 0 1 1 …
$ AGE_C3 : int 13 NA NA NA NA NA NA 12 10 7 …
$ HT_C3 : num 126 NA NA NA NA NA NA 124 128 124 …
$ SEX_C4 : int NA NA NA NA NA NA NA NA NA NA …
$ AGE_C4 : int NA NA NA NA NA NA NA NA NA NA …
$ HT_C4 : int NA NA NA NA NA NA NA NA NA NA …
#No39.Filariasisageデータセット
data(Filariasisage)
str(Filariasisage)
‘data.frame’: 8 obs. of 5 variables:
$ Age_Group : Factor w/ 8 levels “0-9″,”10-19”,..: 1 2 3 4 5 6 7 8
$ Examined : int 247 754 204 146 110 73 73 31
$ Infected : int 11 76 45 38 37 41 46 20
$ Onchocerca_volvulus: int 7 54 32 30 29 34 40 18
$ Other : int 4 22 13 8 8 7 6 2
#No40.FilariasisSexデータセット
data(FilariasisSex)
str(FilariasisSex)
‘data.frame’: 13 obs. of 5 variables:
$ Community : Factor w/ 13 levels “C1″,”C10″,”C11”,..: 1 6 7 8 9 10 11 12 13 2 …
$ Males_Examined : int 120 54 42 103 39 14 319 29 80 173 …
$ Males_Infected : int 16 14 11 17 8 7 55 12 13 21 …
$ Females_Examined: int 133 44 38 57 51 13 110 37 0 14 …
$ Females_Infected: int 19 4 6 6 22 3 9 12 0 0 …
#No41.Filariasistypeデータセット
data(Filariasistype)
str(Filariasistype)
‘data.frame’: 13 obs. of 5 variables:
$ Community : Factor w/ 13 levels “C1″,”C10″,”C11”,..: 1 6 7 8 9 10 11 12 13 2 …
$ Examined : int 253 98 80 150 90 27 429 66 80 187 …
$ Infected : int 35 18 17 23 30 10 64 24 13 21 …
$ Onchocerca_volvulus: int 26 15 15 18 21 7 50 15 12 17 …
$ Others : int 9 3 2 5 9 3 14 9 1 4 …
#No42.Fishデータセット
data(Fish)
str(Fish)
‘data.frame’: 24 obs. of 2 variables:
$ BKT: num 255 1085 969 0 0 …
$ YSC: int 0 0 1 1 0 0 0 0 0 0 …
#No43.fishtoxinデータセット
data(fishtoxin)
str(fishtoxin)
‘data.frame’: 10 obs. of 6 variables:
$ Dose : num 0.01 0.01 0.025 0.025 0.05 0.05 0.1 0.1 0.25 0.25
$ Alfatoxin: Factor w/ 2 levels “total count”,..: 2 1 2 1 2 1 2 1 2 1
$ Tank_1 : int 9 87 30 86 54 89 71 88 66 86
$ Tank_2 : int 5 86 41 86 53 86 73 89 75 82
$ Tank_3 : int 2 89 27 86 64 90 65 88 72 81
$ Tank_4 : int 9 85 34 88 55 88 72 90 73 89
#No44.Frogfoodデータセット
data(Frogfood)
str(Frogfood)
‘data.frame’: 7 obs. of 6 variables:
$ Age : Factor w/ 7 levels “1”,”2″,”3″,”4″,..: 1 2 3 4 5 6 7
$ Body_Weight : int 80 125 175 250 350 450 750
$ Intake_Crabs : num 1.07 7.45 10.43 10.68 14.7 …
$ Intake_Insects: num 1.7 0.96 1.35 1.68 1.19 1.67 0
$ Intake_Larvae : num 1.13 0.34 0.47 0.48 1.37 0 0
$ Total_intake : num 3.9 8.75 12.25 12.84 17.26 …
#No45.Frogmatingデータセット
data(Frogmating)
str(Frogmating)
‘data.frame’: 38 obs. of 2 variables:
$ Bode_Size: int 144 150 144 154 132 148 143 144 146 134 …
$ Mates : int 3 2 1 1 1 1 1 1 1 1 …
#No46.Frog_survivalデータセット
data(Frog_survival)
str(Frog_survival)
‘data.frame’: 8 obs. of 2 variables:
$ Age : int 1 2 3 4 5 6 7 8
$ Individuals: int 9093 35 30 28 12 8 5 2
#No47.GDSデータセット
data(GDS)
str(GDS)
‘data.frame’: 53 obs. of 5 variables:
$ Year : int 1950 1951 1952 1953 1954 1955 1956 1957 1958 1959 …
$ Household_Sector : int 612 583 637 665 719 1046 1178 997 1016 1301 …
$ Private_Corporate_Sector: int 93 136 64 90 118 134 155 121 140 185 …
$ Public_Sector : int 182 266 160 143 169 190 251 266 251 262 …
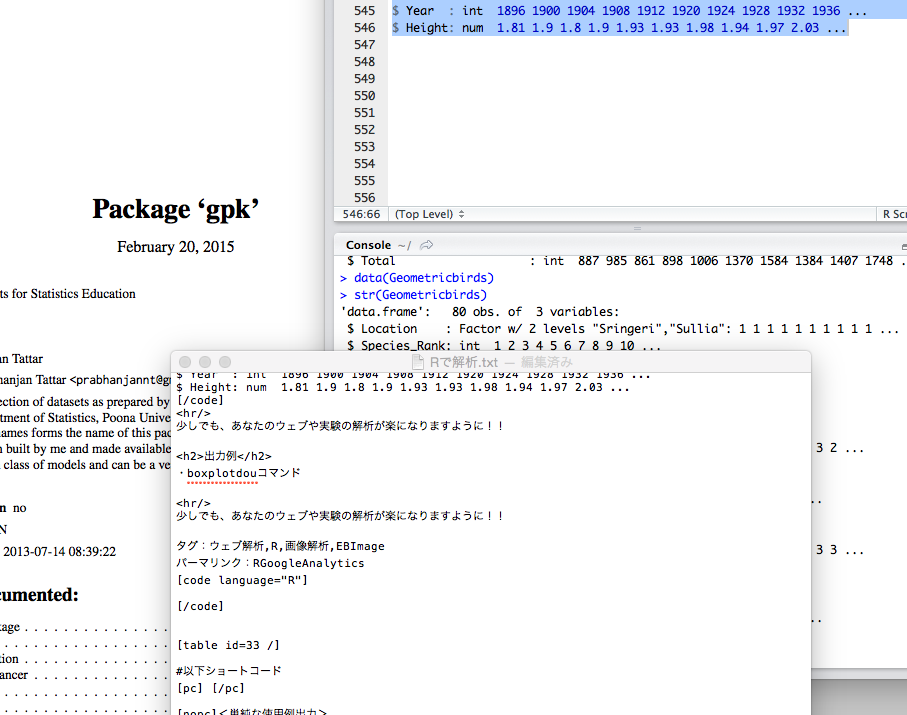
$ Total : int 887 985 861 898 1006 1370 1584 1384 1407 1748 …
#No48.Geometricbirdsデータセット
data(Geometricbirds)
str(Geometricbirds)
‘data.frame’: 80 obs. of 3 variables:
$ Location : Factor w/ 2 levels “Sringeri”,”Sullia”: 1 1 1 1 1 1 1 1 1 1 …
$ Species_Rank: int 1 2 3 4 5 6 7 8 9 10 …
$ Abundance : int 360 205 185 176 173 145 142 136 127 113 …
#No49.Heartデータセット
data(Heart)
str(Heart)
‘data.frame’: 205 obs. of 8 variables:
$ AGE : int 48 36 52 57 35 34 48 68 47 58 …
$ SEX : Factor w/ 3 levels “”,”F”,”M”: 3 3 3 3 3 3 3 3 3 2 …
$ DIABETES : int 0 0 0 0 0 0 0 0 0 1 …
$ HYPERTENSION : int 0 0 0 0 0 0 0 1 1 1 …
$ LDL : int 190 145 183 154 206 102 129 208 145 128 …
$ HDL : int 35 32 50 30 38 47 32 39 26 31 …
$ Primary_Response: int 0 0 0 1 0 0 0 0 1 0 …
$ Drug : Factor w/ 3 levels “”,”P”,”T”: 3 3 3 3 3 3 3 3 3 3 …
#No50.Highjumpデータセット
data(Highjump)
str(Highjump)
‘data.frame’: 24 obs. of 2 variables:
$ Year : int 1896 1900 1904 1908 1912 1920 1924 1928 1932 1936 …
$ Height: num 1.81 1.9 1.8 1.9 1.93 1.93 1.98 1.94 1.97 2.03 …
[/code]
少しでも、あなたのウェブや実験の解析が楽になりますように!!