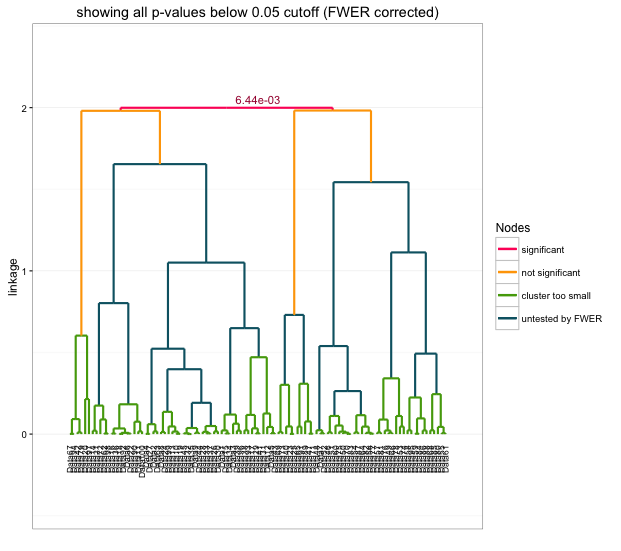
クラスタリング解析に大変便利なパッケージです。Windows環境ではインストールが易かったですが、MACは面倒でした。でも、MAC環境の方にも面倒を乗り越えてぜひ利用してほしいパッケージです。MACでのインストール方法を合わせて紹介します。
パッケージバージョンは0.99.11。実行コマンドはwindows 7およびOS X 10.11.2のR version 3.2.3で確認しています。
Macでの事前準備
下記コマンドを「アプリケーション」、「ユーティリティ」フォルダから「ターミナル」を実行して下記コマンドを実行してください。Xcodeが必要です。もし、XCodeが未インストールの場合はApp Storeよりインストールしてください。
また、macportsのインストールも必要です。下記URLより使用中のmac OSのバージョンに合わせてインストールしてください。
The MacPorts Project -- Download & Installation
The MacPorts Project is an open-source community initiative to design an easy-to-use system for comp...
sudo xcodebuild -license sudo port install gcc48 g++-mp-4.8 -v mkdir ~/.R nano ~/.R/Makevars CXX=g++-mp-4.8 -arch x86_64 CC=gcc-mp-4.8 SHLIB_OPENMP_CXXFLAGS= -fopenmp #上記実行後"controlキー + o",そして"return"キー,"controlキー + x"を実行
パッケージのインストール
下記、コマンドを実行してください。本パッケージの利用は依存パッケージがかなりシビアです。もし、「devtools::install_github(“pkimes/sigclust2”)」コマンド実行後にエラーが出る場合は、エラーメッセージで表示されるパッケージをインストールしてください。
#念のため実行
source("https://bioconductor.org/biocLite.R")
biocLite(c("GO.db", "preprocessCore", "preprocessCore"))
#MACは以下を実行
install.packages("Matrix")
install.packages(c("Rcpp", "RcppEigen", "Rclusterpp"), type = "source")
#パッケージのインストール
install.packages("devtools")
devtools::install_github("pkimes/sigclust2")
実行コマンド
詳細はコメント、パッケージのヘルプを確認してください。
#パッケージの読み込み
library(“sigclust2”)
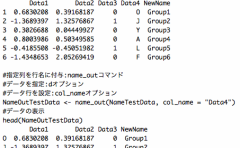
###データ例の作成#####
n
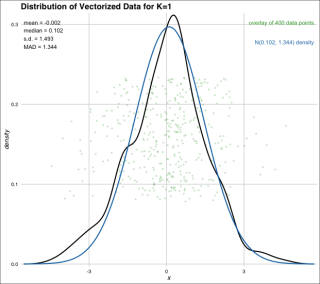
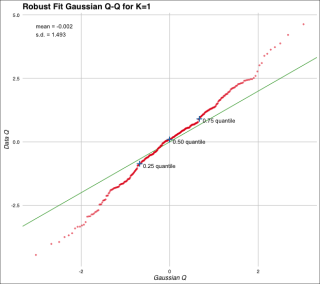
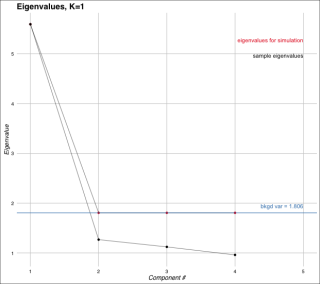
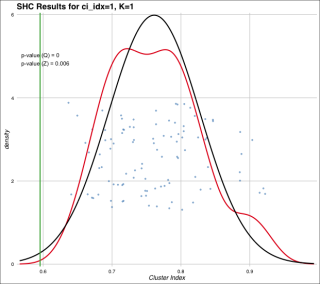
出力例
少しでも、あなたのウェブや実験の解析が楽になりますように!!