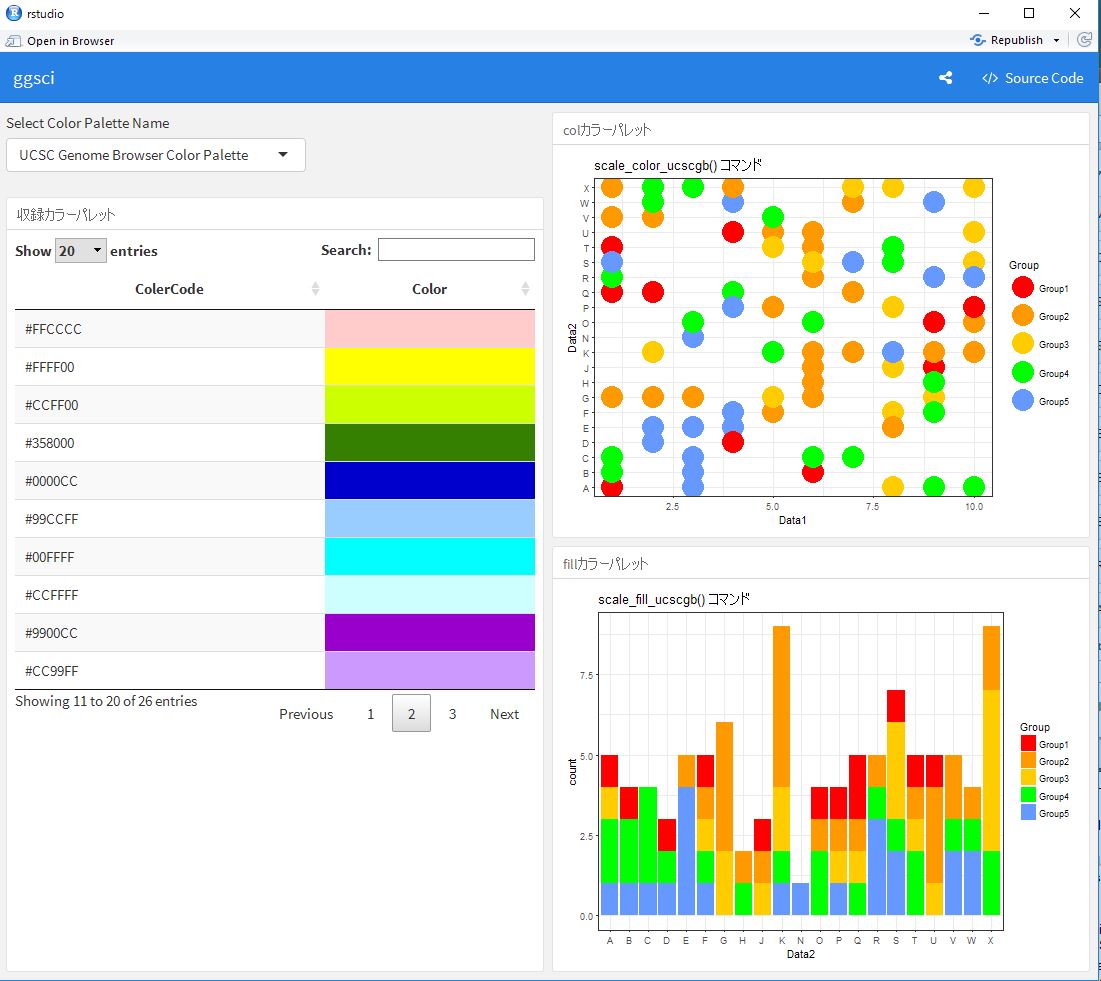
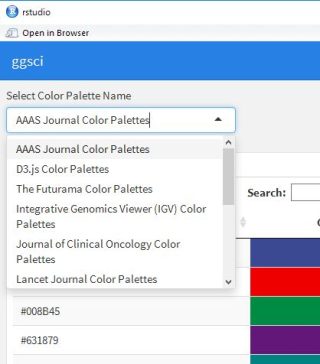
ggplot2でも利用できるカラーパレットが収録されているパッケージの紹介です。コマンドの紹介のRmdファイルを実行するとカラーパレットを確認できるようにしました。
パッケージバージョンは2.9。実行コマンドはwindows 11のR version 4.1.2で確認しています。
パッケージのインストール
下記コマンドを実行してください。
#パッケージのインストール
install.packages("ggsci")コマンドの紹介
詳細はコマンド、各パッケージのヘルプを確認してください。以下からファイルをダウンロードしRStudioで実行しても同じコマンドの紹介内容と同じ結果が得られます。なお、拡張子はRmdです。
</p>---
title: "ggsci"
output:
flexdashboard::flex_dashboard:
orientation: columns
social: menu
source_code: embed
runtime: shiny
---
```{r global, include=FALSE}
#必要パッケージの読み込み
if(!require("ggsci", quietly = TRUE)){
install.packages("ggsci");require("ggsci")
}
if(!require("tidyverse", quietly = TRUE)){
install.packages("tidyverse");require("tidyverse")
}
if(!require("flexdashboard", quietly = TRUE)){
install.packages("flexdashboard");require("flexdashboard")
}
if(!require("DT", quietly = TRUE)){
install.packages("DT");require("DT")
}
###データ例の作成#####
n <- 100
TestData <- data.frame("Group" = sample(paste0("Group", 1:5), n, replace = TRUE),
"Data1" = sample(1:10, n, replace = TRUE),
"Data2" = sample(LETTERS[1:24], n, replace = TRUE))
########
#「ggsci」パッケージ:カラーパレット名
GgSColName <- c("AAAS Journal Color Palettes", "D3.js Color Palettes", "The Futurama Color Palettes",
"Integrative Genomics Viewer (IGV) Color Palettes", "Journal of Clinical Oncology Color Palettes",
"Lancet Journal Color Palettes", "LocusZoom Color Palette", "NEJM Color Palettes",
"NPG Journal Color Palettes", "Rick and Morty Color Palettes", "The Simpsons Color Palettes",
"Star Trek Color Palettes", "The University of Chicago Color Palettes",
"UCSC Genome Browser Color Palette")
#「ggsci」パッケージ:ggplot2で使えるシンボル色コマンド
GgScalCol <- c("scale_color_aaas()", "scale_color_d3()", "scale_color_futurama()",
"scale_color_igv()", "scale_color_jco()", "scale_color_lancet()",
"scale_color_locuszoom()", "scale_color_nejm()", "scale_color_npg()",
"scale_color_rickandmorty()", "scale_color_simpsons()", "scale_color_startrek()",
"scale_color_uchicago()", "scale_color_ucscgb()")
#「ggsci」パッケージ:ggplot2で使える塗色コマンド
GgScalFill <- c("scale_fill_aaas()", "scale_fill_d3()", "scale_fill_futurama()",
"scale_fill_igv()", "scale_fill_jco()", "scale_fill_lancet()",
"scale_fill_locuszoom()", "scale_fill_nejm()", "scale_fill_npg()",
"scale_fill_rickandmorty()", "scale_fill_simpsons()", "scale_fill_startrek()",
"scale_fill_uchicago()", "scale_fill_ucscgb()")
#「ggsci」パッケージ:収録カラーパレットコマンド
GgScalPal <- c("pal_aaas()", "pal_d3()", "pal_futurama()",
"pal_igv()", "pal_jco()", "pal_lancet()",
"pal_locuszoom()", "pal_nejm()", "pal_npg()",
"pal_rickandmorty()", "pal_simpsons()", "pal_startrek()",
"pal_uchicago()", "pal_ucscgb()")
#「ggsci」パッケージ:収録各カラーパレットの色数
GgScalNO <- c(10, 10, 12, 51, 10, 9, 7, 8, 10, 12, 16, 7, 9, 26)
```
Column {data-width=500}
-------------------------------------
```{r}
selectInput('Colpal', 'Select Color Palette Name', GgSColName)
```
###収録カラーパレット
```{r}
renderDataTable({
ColCodeData <- eval(parse(text = paste0(GgScalPal[which(GgSColName == input$Colpal)], "(", GgScalNO[which(GgSColName == input$Colpal)], ")")))
ColCodeData <- data.frame("ColerCode" = strtrim(ColCodeData, 7),
"Color" = rep("", length(ColCodeData)))
datatable(ColCodeData, rownames = FALSE,
options = list(pageLength = 10, lengthMenu = c(20, 40, 100))) %>%
formatStyle("Color", valueColumns = "ColerCode",
backgroundColor = styleEqual(ColCodeData[, 1], ColCodeData[, 1]))
})
```
Column {data-width=500}
-------------------------------------
###colカラーパレット
```{r}
renderPlot({
ggplot(TestData,
aes(x = Data1, y = Data2, col = Group)) +
geom_point(size = 10) +
theme_bw() +
eval(parse(text = GgScalCol[which(GgSColName == input$Colpal)])) +
labs(title = paste0(GgScalCol[which(GgSColName == input$Colpal)], " コマンド"))
})
```
###fillカラーパレット
```{r}
renderPlot({
ggplot(TestData,
aes(x = Data2, fill = Group)) +
geom_histogram(stat = "count") +
theme_bw() +
eval(parse(text = GgScalFill[which(GgSColName == input$Colpal)])) +
labs(title = paste0(GgScalFill[which(GgSColName == input$Colpal)], " コマンド"))
})
```出力例
少しでも、あなたの解析が楽になりますように!!