生存曲線の作成が大変簡単なパッケージの紹介です。Log-rank検定の結果や生存数のテーブルなどの同時プロットが可能です。非常に簡単で見栄えの良いグラフが作成可能です。
パッケージバージョンは0.4.9。実行コマンドはwindows 11のR version 4.1.2で動作を確認しています。
パッケージのインストール
下記コマンドを実行してください。
#パッケージのインストール
install.packages("survminer")コマンドの紹介
詳細はコマンド、パッケージのヘルプを確認してください。
#パッケージの読み込み
library("survminer")
###データ例の作成#####
#Status:0=alive,1=deadを示す
#自身のデータも生存、死亡を数字に変換してください
n <- 150
set.seed(1234)
TestData <- data.frame(ID = 1:n, Group = rep(c("A", "B"), length = n),
Day = sample(0:20, n, replace = TRUE),
Status = sample(0:1, n, replace = TRUE))
#survivalパッケージのSurv,survfitコマンドで生存計算
library("survival")
ServResult <- survfit(Surv(Day, Status) ~ Group, data = TestData)
######## #生存曲線の作成:ggsurvplotコマンド
#Surv,survfitコマンドの結果を指定:fitオプション
#Log-rank検定結果を表示:pvalオプション
#生存時間(x軸)の分割数を設定:break.time.byオプション
#信頼区間を表示:conf.intオプション
#信頼区間の表示設定:conf.int.styleオプション;"ribbon"or"step"
#打切り数を棒グラフで表示:ncensor.plotオプション
#50%生存の補助線を表示:surv.median.lineオプション:
#"none","hv"(水平垂直に追加),"h"(水平に追加),"v"(垂直に追加)
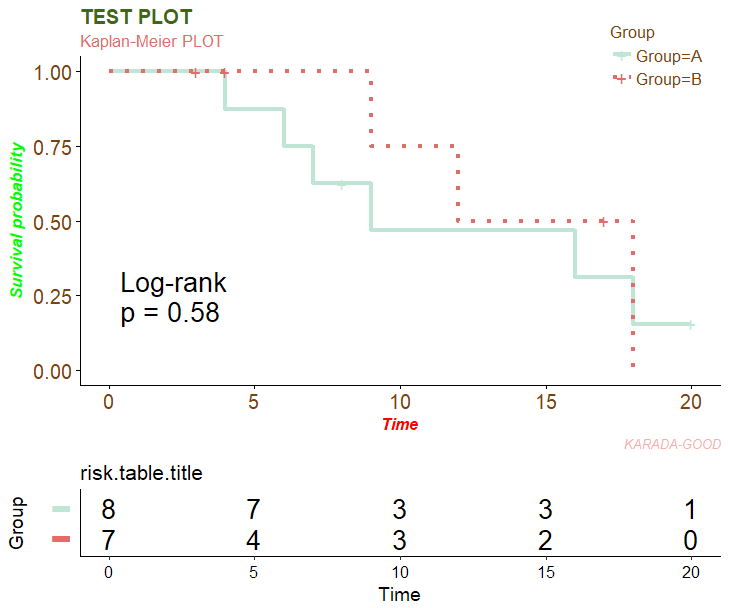
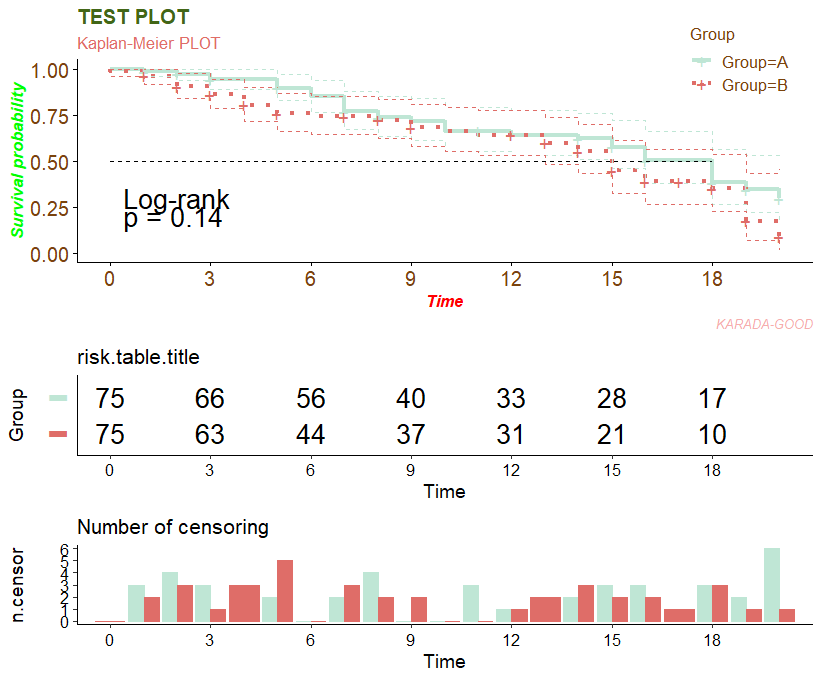
ggsurvplot(fit = ServResult, data = TestData,
#Kaplan-Meier PLOTの表示内容を指定
palette = c("#bfe6d5", "#df6d68"), pval = TRUE, break.time.by = 3,
pval.method = TRUE, risk.table = TRUE,
conf.int = TRUE, conf.int.style = "step",
ncensor.plot = TRUE, size = 1.5, linetype = c(1, 3),
surv.median.line = "h",
#Kaplan-Meier PLOT部分の体裁を調整
title = "TEST PLOT", subtitle = "Kaplan-Meier PLOT",
caption = "KARADA-GOOD", legend.title = "Group",
font.title = c(15, "bold", "#426617"),
font.subtitle = c(13, "plain", "#df6d68"),
font.caption = c(10, "italic", "#f6adad"),
font.x = c(13, "bold.italic", "red"),
font.y = c(13, "bold.italic", "green"),
font.tickslab = c(15, "plain", "#7a430e"),
legend = c(0.9, 1), font.legend = c(13, "plain", "#7a430e"),
pval.size = 7,
#"1","n","sqrtN","S1","S2","FH"が選択可能
###それぞれの内容#####
#1;log-rank, n;Gehan-Breslow (generalized Wilcoxon)
#sqrtN;Tarone-Ware, S1;Peto-Peto's modified survival estimate
#S2;modified Peto-Peto (by Andersen), FH ;Fleming-Harrington(p=1, q=1)
########
log.rank.weights = "1",
#risk.table部分の体裁を調整
risk.table.title = "risk.table.title", risk.table.y.text.col = TRUE,
risk.table.y.text = FALSE, fontsize = 7)出力例
少しでも、あなたの解析が楽になりますように!!